Home > Pymol > 保存度による色分け
The Consurf Serverを使ったアミノ酸の保存度による色分けの方法です。ここでは5つ以上の配列を使います。 4つ以下だとThe Consurf Serverでは計算してもらえません。4つ以下の場合はコチラをご参照ください。Multi sequence aligmentファイルの作成
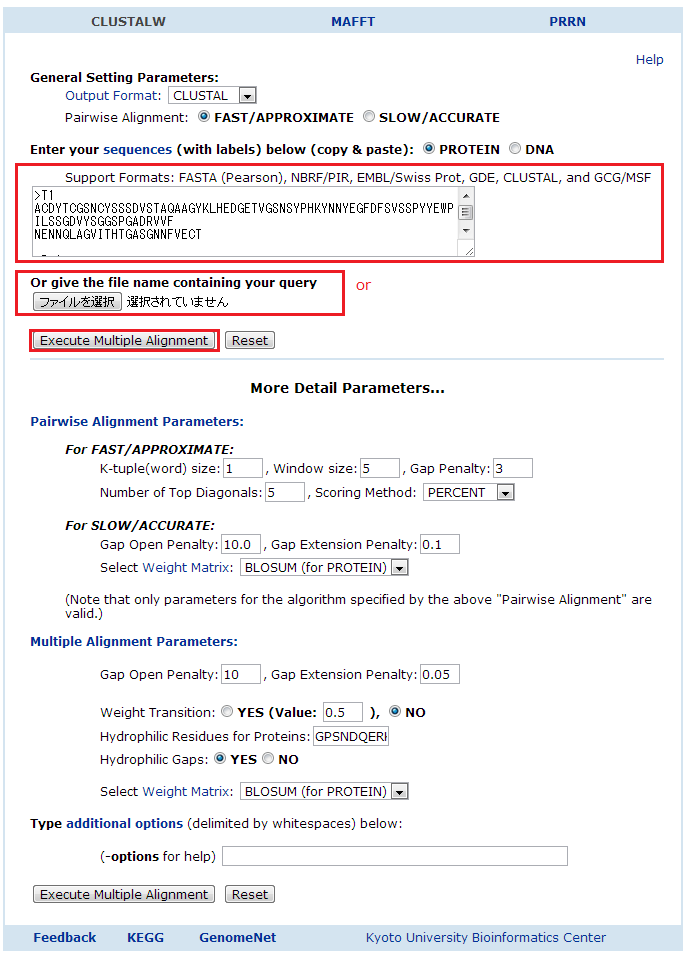
- FASTAファイルのアップロード CLUSTALWで比較したいアミノ酸配列をまとめたFASTAをアップロードするか、 テキストを貼り付けます。
- Execute Multiple Aligmentをクリック
- clustalw.alnを保存
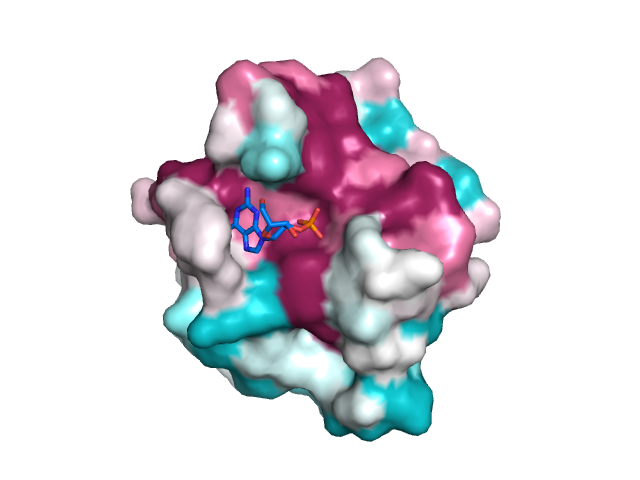
 例ではT1タイプのRNase、T1/Po1/He1/Ms/F1の5つの保存性をT1(PDB ID:2B4U)に色をつけます。
例ではT1タイプのRNase、T1/Po1/He1/Ms/F1の5つの保存性をT1(PDB ID:2B4U)に色をつけます。今回使用したFASTAファイル
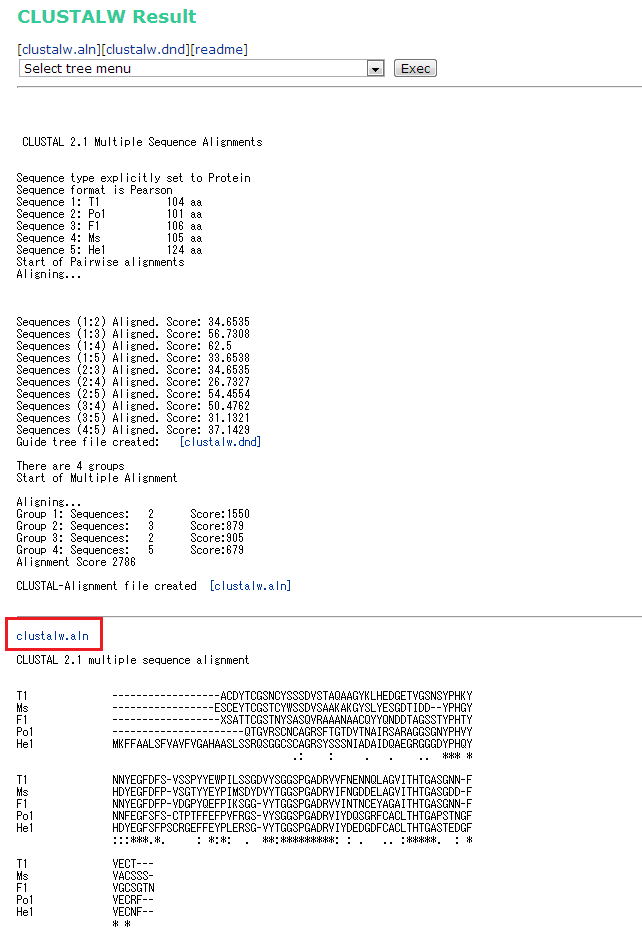 今回使用したアライメントファイル
今回使用したアライメントファイル
The ConSurf Server
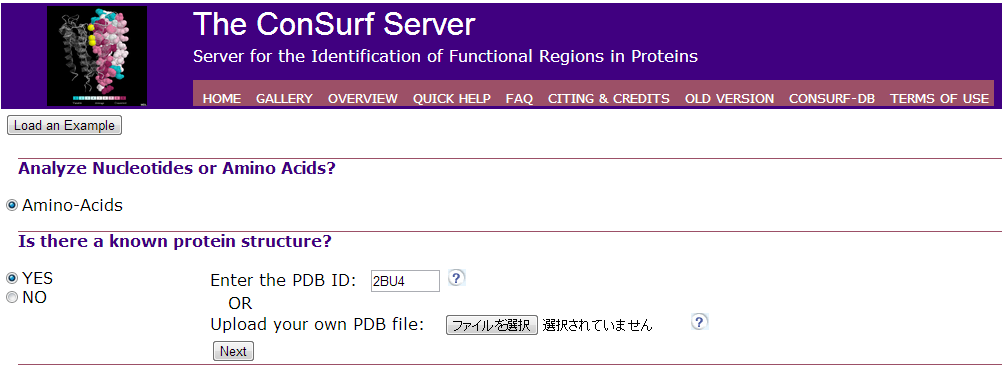
The ConSurf Serverにアクセス- Analyze Nucleotides or Amino Acids? →Amino-Acids
- Is there a known protein structure? →YRS
- Do you have a Multiple Sequence Aligment(MSA) to upload? →アライメントファイル(○○.aln)をアップロード
- Do you have a tree file to upload? →No
- Job titleに任意の名前とE-Mailを入力して、submitをクリック
- 計算が終わったら、Follow the instructions to produce a PyMol figure (For users of PyMol)をクリック
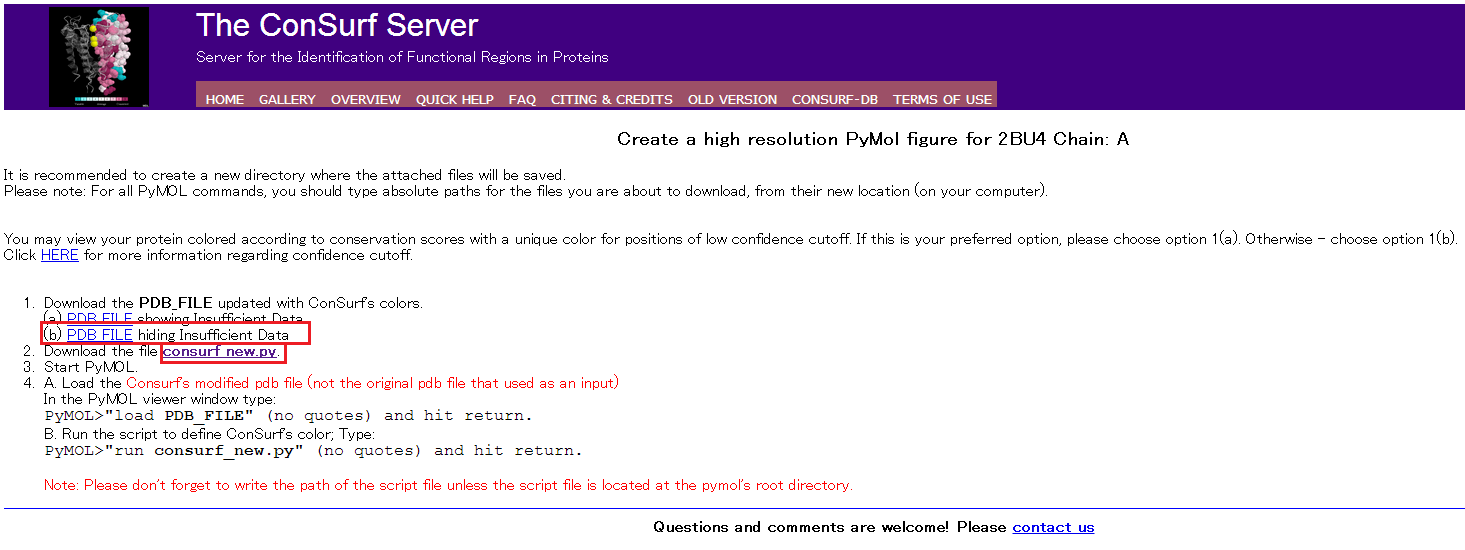
- PDBファイルとPythonスクリプトのダウンロード
- (b) PDB_FILE hiding Insufficient DataをクリックしてPDBファイルをダウンロード
- consurf_new.pyをダウンロード
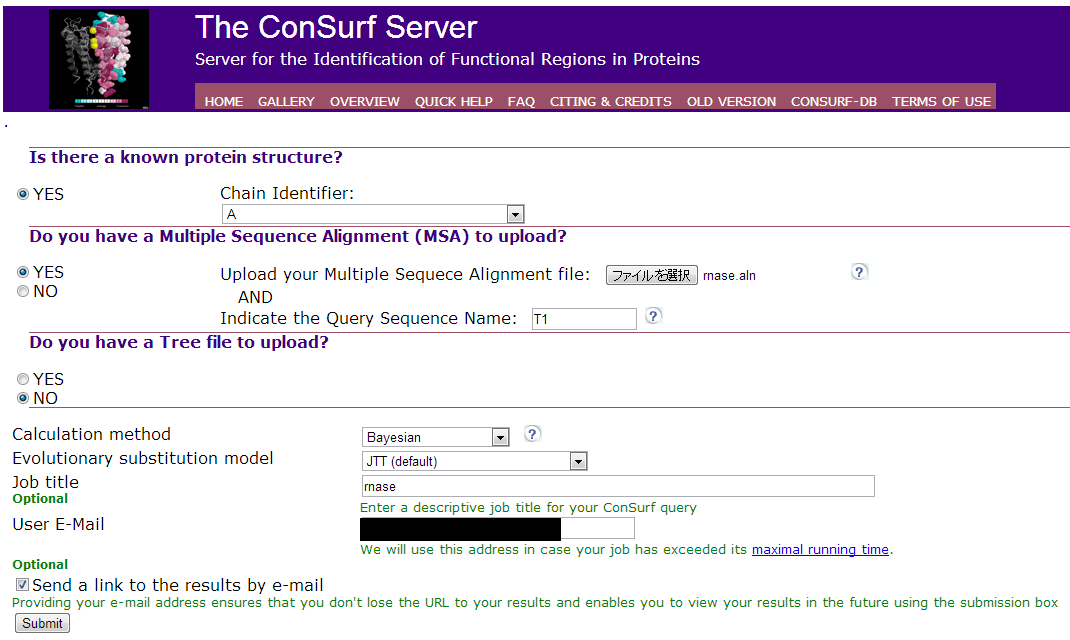
→PDB IDを入力 or PDBファイルをアップロード
"next" をクリック
→Indicate the Query Sequence NameにPDBファイルに対応する配列名を入力 (PDB ID:2B4Uの配列はアライメントファイルのT1に相当するので、今回はT1と入力)

PyMOLで色をつける
- PyMOLでPDBファイルを開く
- Run → consurf_new.pyを開く 分子表面を表示して完成です。保存性の高い場所が紫、低いところが水色に色分けされます。