ACHIEVEMENTS
![]()
ACHIEVEMENTS研究成果
-

- ACHIEVEMENTS
- 細胞の多面的な遺伝子解析ソフトウェアASURATを開発 ―専門家の経験に依存しない”機能アノテーション”を用いた細胞分類法―
プレスリリース
2022.09.07
細胞の多面的な遺伝子解析ソフトウェアASURATを開発 ―専門家の経験に依存しない”機能アノテーション”を用いた細胞分類法―
研究成果のポイント
- ・遺伝子発現情報から細胞分類と生物学的解釈を同時に実現するソフトウェアASURATを開発
- ・遺伝子の機能アノテーション情報を用いた細胞分類法の開発により、遺伝子発現量に主眼を置く従来のデータ解析手法では見落とされてきた生体組織の悪性化部位を検出
- ・細胞の機能異常を詳細に検査するシステムの開発により疾患診断の精度向上に貢献
概要
大阪大学蛋白質研究所の飯田 渓太 助教、Johannes Nicolaus Wibisana 博士前期課程学生(現 沖縄科学技術大学院大学 博士後期課程学生)、岡田 眞里子 教授らは、京都大学大学院医学研究科の井上 正宏 特定教授、近藤純平 特定助教(現 大阪大学大学院医学系研究科 准教授)との共同研究において細胞の機能別分類を実現する遺伝子発現解析ソフトウェアASURAT(functional annotation-driven unsupervised clustering of single-cell transcriptomes)を開発し、敗血症、肺がん、膵がんなどの患者データから疾患に関わる細胞集団とその生物機能(代謝異常や薬剤耐性など)を新たに見出すことに成功しました。
遺伝子発現量に主眼を置く従来のデータ解析手法では、細胞内で活性化している遺伝子による細胞分類が行われてきました。しかし、こうした方針のみでは同じ細胞型であっても発現量が細胞ごとに大きく異なるような遺伝子発現パターンを解析することは困難でした。そこで、本研究では細胞種別、生化学反応、分子パスウェイなどに関する遺伝子の機能アノテーション※1の情報を用いて細胞を多面的に分類するソフトウェアASURATを開発しました(図1)。応用例として、敗血症患者の1細胞トランスクリプトームデータ※2から病態の進行に伴い減少するマクロファージの亜集団を新規に発見し、それらが他のマクロファージとは異なる代謝状態にあることを見出しました。また、膵がん患者の1細胞トランスクリプトームデータと空間トランスクリプトームデータ※3から、がんの悪性化に関わる細胞集団とその機能的特徴(蛋白質分解能など)を予測し、その結果を組織画像との比較により検証しました。
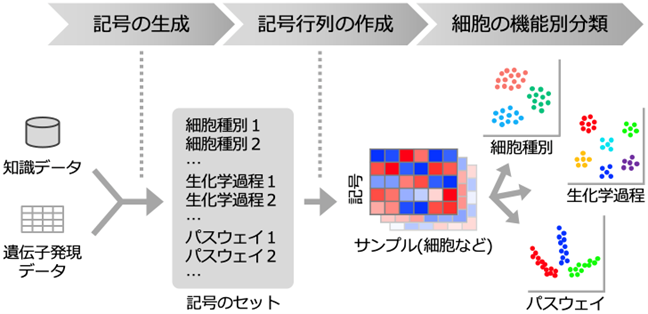
図1 ASURATが可能にする細胞の多面的な機能別分類の概要。
入力した知識データと遺伝子発現データをもとに記号と呼ばれる生物機能に関する変数セットを生成する。
記号の解析により細胞分類と生物学的解釈が同時に実現可能となる。
研究の背景
細胞内のRNA分子の量(遺伝子発現量)を網羅的に計測することのできる1細胞トランスクリプトーム解析の実験技術の精度は最近10年間で飛躍的に向上しました。特に、近年は細胞の位置情報と発現情報を同時に取得することのできる空間トランスクリプトーム解析が大きな注目を集めています。その結果、生体組織に潜むがん細胞などを分子レベルで同定することが可能になりました。しかし、遺伝子発現量に主眼を置く従来のデータ解析手法では、細胞内でどのような生物機能が働いているかまでは推定することが困難でした。遺伝子は非常に多くの生物機能に関わるため、どれが真に重要な機能であるかを判別する方法が存在しなかったためです。そのため、これまでは研究者の専門知識と経験による細胞の機能推定が行われてきました。しかし、こうした方針は網羅性に乏しく、特に疾患遺伝子マーカーが明瞭でない難治性がんなどでは悪性細胞の機能的特徴(薬剤耐性や浸潤能など)の解明が大きな課題となっています。そこで、本研究では遺伝子の機能アノテーション情報を用いて細胞を機能別に分類するソフトウェアの開発を目指しました。
研究の内容
我々が着目したのは細胞種別、生化学反応、パスウェイなどに関する遺伝子情報を集約した公共のデータベースです。本研究ではこれらのデータベースと遺伝子発現データから生物機能のセットを生成する方法の開発に取り組み、多様な機能アノテーション情報によって細胞を機能別に分類するソフトウェアASURATを開発しました(図1)。これにより、従来の遺伝子ベースの解析では見落とされてきた細胞集団を新たに発見することが可能になります。応用例として、膵がん患者の1細胞トランスクリプトームデータと空間トランスクリプトームデータから膵管腺癌の亜集団とその機能的特徴(蛋白質分解能など)を新たに予測し、その結果を組織画像との比較により検証することに成功しました(図2)。現在、こうした複合的なデータセットに適用可能な解析ツールは限られており、従来法は適用が困難です。こうした中、ASURATはいずれのデータにも適用可能な本邦発の革新的解析ツールとして期待されます。ASURATは応用範囲が広く、データベースの使い方を工夫することで幅広い生命データの解析が可能になると期待されます。
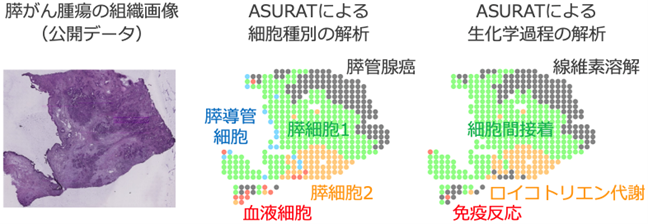
図2 ASURATによる膵がん腫瘍のデータ解析例。細胞種別だけでなく、生化学過程やパスウェイなどの細胞機能を網羅的に推定することが可能。
公開データの出典:Moncada et al, Nat. Biotechnol. 38, 333-342 (2020)。
本研究成果が社会に与える影響(本研究成果の意義)
本研究により組織画像の観察のみからは判別することの難しい細胞内の生物機能をこれまで以上に詳細かつ網羅的に解明できる可能性が示唆されました。今後、薬剤投与やゲノム変異などが引き起こす生物機能の情報データベースをASURATに入力することが可能になれば、生体組織の機能異常をより精確に検出し治療効果を予測するなど、創薬の研究分野にも貢献できることが期待できます。
特記事項
本研究成果は国際科学誌『Bioinformatics』より8月5日(金)午前1時(日本時間)に公開されました。また、本研究において開発した遺伝子発現解析ソフトウェアASURATは査読付きのオープンソースのソフトウェアとしてBioconductorに公開されました(DOI: 10.18129/B9.bioc.ASURAT)。
タイトル:“ASURAT: functional annotation-driven unsupervised clustering of single-cell transcriptomes”
著者:Keita Iida1,*, Jumpei Kondo2,3, Johannes Nicolaus Wibisana1, Masahiro Inoue3, Mariko Okada1
所属:1:大阪大学蛋白質研究所、2:大阪大学大学院医学系研究科、3:京都大学医学研究科、*:責任著者
掲載雑誌:Bioinformatics
DOI:https://doi.org/10.1093/bioinformatics/btac541
タイトル:“ASURAT: Functional annotation-driven unsupervised clustering for single-cell data”
著者:Keita Iida
掲載元:Bioconductor
DOI:10.18129/B9.bioc.ASURAT
本研究は日本学術振興会(JSPS)科学研究費補助金 若手研究「一細胞データから遺伝子の制御構造を定量推定するための確率・統計理論の構築」(研究代表者:飯田 渓太、20K14361)、蛋白質研究所新分野開拓支援プログラム「がん可塑性の機構解明に向けた知識データ活用型の革新的オミクス解析」(研究代表者:飯田 渓太)、科学技術振興機構(JST)ムーンショット型研究開発事業「オミクスデータの記号学的分類を可能にする数理研究」(研究代表者:飯田 渓太、JPMJMS2021)、国立研究開発法人日本医療研究開発機構(AMED)次世代がん医療創生研究事業(研究代表者:野田 哲生)、JST CREST バイオDX領域「自然言語処理とシミュレーションによる細胞制御探索法の構築」(研究代表者:岡田 眞里子、JPMJCR21N3)などの支援の下に行われました。
用語説明
※1 遺伝子の機能アノテーション:
生命科学分野ではヒトやマウスを含むさまざまな生物種を用いた実験と解析により、多くの遺伝子とその機能が明らかにされてきました。これらの知見はデータベース化されており公共利用が可能です。遺伝子の機能アノテーションとは、こうしたデータベースを用いて研究対象とする遺伝子セットに生物機能を割り当てる作業のことです。例えば、KEGG(Kyoto Encyclopedia of Genes and Genomes)のパスウェイデータベースには340個の分子パスウェイとそれに関連する遺伝子セットの情報が登録されており(2020年12月時点)、パスウェイに関する機能アノテーションを行うことが可能です。本研究ではこの他にも細胞種別、疾患種別、生化学反応に関するデータベースを取得し、ユーザーが使用しやすい形のテーブルデータとして整理しました。
※2 1細胞トランスクリプトームデータ:
ヒトの細胞には蛋白質をコードした遺伝子が約20,000個存在しており(Salzberg, BMC Biol., 2018)、この領域から生命維持に必要なRNA分子が産生されています。1細胞トランスクリプトームとは1細胞レベルで得られた細胞内の全RNA分子の情報を指します。次世代シーケンサを用いた細胞内分子の計測技術は最近の10年間で飛躍的に向上しており、今日では一度のサンプル回収で数百万の細胞に対する1細胞トランスクリプトームデータを取得することが可能になっています。
※3 空間トランスクリプトームデータ:
先行研究(Moncada et al., 2020)では、スライドガラス上に格子状に整列したマイクロアレイのスポットに膵がん腫瘍の組織切片を貼り付ける方法により、腫瘍に存在する細胞のRNA情報と位置情報を同時にもつ空間トランスクリプトームデータが取得されました。ただし、ここで得られた空間トランスクリプトームは1スポットあたり20-70細胞が平均化された粗いデータであり、1細胞レベルではありません。そこで先行研究(Moncada et al., 2020)では、同じ組織から1細胞トランスクリプトームデータを取得し、これらのデータを統合することでスポット間でのバイアスの少ない細胞分類を可能にしました。
飯田 渓太 助教のコメント
| 問題設定からデータ収集、理論構築を経てソフトウェアの作成に至る、どの過程でも苦慮しましたが、共同研究者や多くの方に支えられました。 |
【蛋白質研究所】研究者紹介:岡田 眞里子教授、飯田 渓太助教

左:岡田 眞里子教授 右:飯田 渓太助教
―本研究成果の苦労された点は?―
飯田渓太助教:着想から開発、問題設定からデータ収集、理論構築を経てソフトウェア「ASURAT」を作成し、完成に至るまでのどの過程でも苦慮しました。共同研究者や多くの方に支えられて感謝しております。
岡田教授:理論構築を経てソフトウェアをパブリックにオープンするまでに時間がかかり大変だったという印象です。競争の激しい分野なので、新規性がないとこういったソフトウェアは認めてもらえません。空間トランスクリプトーム解析のソフトウェアは少ないので、多くの方に使って頂けれるとうれしく思います。
―ひと言コメントをお願いいたします―
AMEDの次世代がん医療創生研究事業(研究代表者:野田 哲生先生)の仕事がきっかけとなり、井上先生、近藤先生と出会い、ディスカッションした経緯があって、今回の論文とソフトウェア「ASURAT」の成果となりました。次世代がん医療創生研究事業には感謝です。






