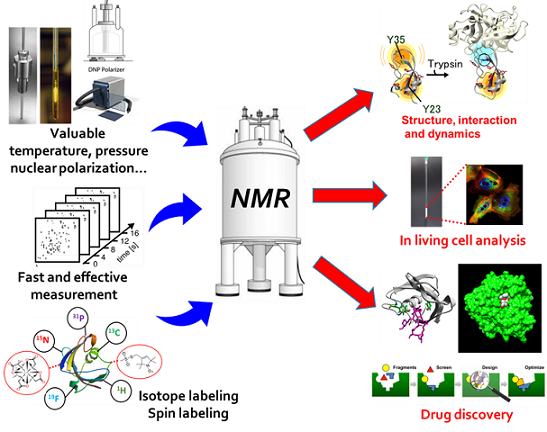
【研究概要】
溶液NMR法は、蛋白質の動的な構造情報を原子分解能で取得できるだけでなく、蛋白質が実際に働く、細胞環境下で解析することができます。これまでにも蛋白質の立体構造や相互作用、運動性解析に利用され、創薬研究等にも応用されています。(下図)
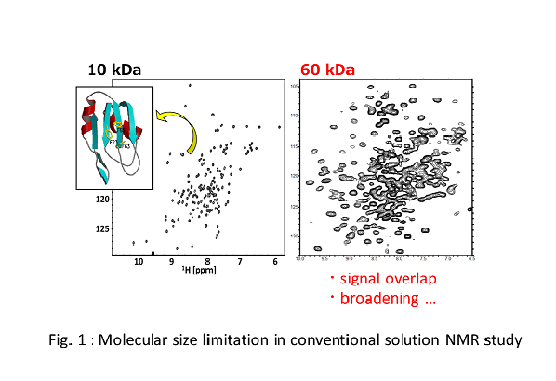
一方で、蛋白質の分子量が増大すると、NMRの信号感度が著しく低下してしまいます(Fig.1) 。 したがって、NMR法が対象とする蛋白質試料は分子量が30 kDa以下のものが大半であり、さまざまな生命機能を詳細に解明するうえでは、いくつかの問題点を克服せねばなりません。
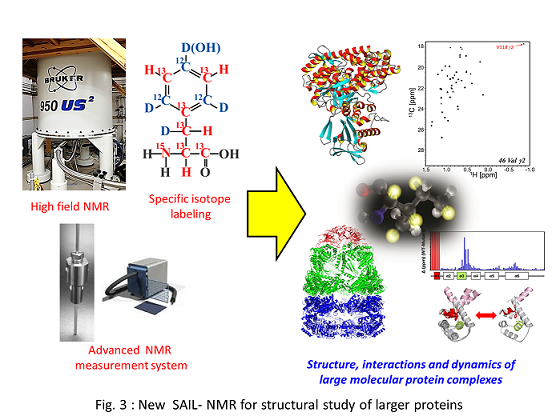
この”分子量の壁”を打破すべく、私たちは高度な安定同位体標識技術を開発してきました。首都大学東京の甲斐荘正恒教授が開発した立体整列同位体標識 (Stereo-array isotope labelling; SAIL )法の出現により、 分子量40 kDa 程度の高分子量蛋白質についても、高精度に立体構造や運動性を解析することが可能となりました(Fig.2)。

現在SAIL法のさらなる改良や高磁場NMR装置を利用した新たな測定法を駆使して、従来よりもさらに高分子量な蛋白質や蛋白質複合体(分子量80 – 1000 kDa)についても、立体構造の決定ならびに相互作用に伴う蛋白質の動態変化を解析する手法の開発を進めています。これらの手法を駆使して、膜蛋白質や抗体蛋白質、べん毛モーターなどの巨大蛋白質複合体について、動態構造を明らかにすることを目指します(Fig.3)。
【Research interest】
Our research is concerned with structural and dynamical aspects of protein function.
We are interested in elucidating correlation between structure,dynamics and biological functions of macromolecular proteins.
To pursueour goals, we use nuclear magnetic resonance spectroscopy (NMR).
The NMR method targets solution proteins under various conditions, included in living cells. Therefore, it allows us to obtain crucial information for
elucidating biological functions of proteins at an atomic resolution. Although
NMR is powerful tool, its potential has been limited due to the presence of signal broadening, overlapping and lower sensitivity in large molecular
weight proteins (ex. > 25 kDa). To overcome this problem, various isotope labeling techniques were developed. Our individual techniques, Stereo array
isotope labeling (SAIL) method contributed significantly to improve NMR spectra of larger proteins and succeeded in determining precise structure of 42 kDa proteins.
For much larger proteins and protein complexes (i.e. > 50 kDa), presently,
we use site specific amide (15NH) and methyl (13CH3) labeling methods.
Although it allows us to obtain highly sensitive methyl and amide TROSY signals that enable the study of local structure, it’s
still not sufficient for NMR-based structure determination which is needed for understanding precise protein function. In order to solve this problem,
we have been developing the new SAIL amino acids. Our new SAIL methodsallow us to observe the several 1H-13C signals even in 80 – 1000 kDa large molecular proteins.
This information is very useful for understanding the precise structure and dynamic property of various macromolecular proteins such as membrane proteins and protein-protein complexes.
Key words
Structural biology, Molecular biology, NMR, Stable isotope labeling, Dynamics
【研究課題 Research theme】
1) SAIL-NMR法を活用した高分子量蛋白質の溶液構造解析法の開発
(Structural studies of larger proteins using new SAIL amino acids)
2) 多様なアミノ酸残基側鎖を対象とした蛋白質の構造動態解析法の開発
(Development of new structural dynamics analysis method using SAIL-NMR)
3) 新しい安定同位体標識蛋白質の調製法の開発
(Development of cost effective protocols to prepare isotope labeled proteins)
4) 高解像度細胞生物学・創薬科学研究に向けたNMR測定法の開発
(New In-cell NMR method for high resolution cell biology and drug discovery)
5) べん毛モーター蛋白質複合体の構造生物学研究
(Integrated structural study for Bacterial flagellar motor machinery)